6. 反応速度式の関数でODEを解く方法¶
odeソルバーはNetworkModelを受け入れますが、
ode.ODESimulatorはそのモデルクラスode.ODENetworkModelと
ode.ODEReactionRuleを機能拡張のために所有しています。
これらのクラスのインタフェースは
ModelクラスとReactionRuleとほとんど同じです。
ここでは、特に
ode.ODERatelawに関するodeに特有の使用法について説明します。
In [1]:
%matplotlib inline
from ecell4 import *
しかし、ode における反応速度式のサポートはまだ開発中の状態です。
一部の関数は、将来廃止される可能性があります。
現在、反応速度式を有効にするには、オプション
ecell4.util.decorator.ENABLE_RATELAWを次のように有効にする必要があります:
In [2]:
util.decorator.ENABLE_RATELAW = True
6.1. ode.ODEReactionRule¶
ode.ODEReactionRuleはReactionRuleとほぼ同じメンバーを持っています。
In [3]:
rr1 = ReactionRule()
rr1.add_reactant(Species("A"))
rr1.add_reactant(Species("B"))
rr1.add_product(Species("C"))
rr1.set_k(1.0)
print(len(rr1.reactants())) # => 2
print(len(rr1.products())) # => 1
print(rr1.k()) # => 1.0
print(rr1.as_string()) # => A+B>C|1
2
1
1.0
A+B>C|1
In [4]:
rr2 = ode.ODEReactionRule()
rr2.add_reactant(Species("A"))
rr2.add_reactant(Species("B"))
rr2.add_product(Species("C"))
rr2.set_k(1.0)
print(len(rr2.reactants())) # => 2
print(len(rr2.products())) # => 1
print(rr2.k()) # => 1.0
print(rr2.as_string()) # => A+B>C|1
2
1
1.0
A+B>C|1
共通のメンバーに加えて、
ode.ODEReactionRuleは各Speciesの化学量論的係数を格納することができます:
In [5]:
rr2 = ode.ODEReactionRule()
rr2.add_reactant(Species("A"), 1.0)
rr2.add_reactant(Species("B"), 2.0)
rr2.add_product(Species("C"), 2.5)
rr2.set_k(1.0)
print(rr2.as_string())
A+2*B>2.5*C|1
次のように係数にアクセスすることもできます。
In [6]:
print(rr2.reactants_coefficients()) # => [1.0, 2.0]
print(rr2.products_coefficients()) # => [2.5]
[1.0, 2.0]
[2.5]
6.2. ode.ODERatelaw¶
ode.ODEReactionRuleはode.ODERatelawにバインドされます。
ode.ODERatelawは与えられたSpeciesの値に基づいて導関数(フラックスまたは速度)を計算する関数を提供します。
ode.ODERatelawMassActionはode.ODEReactionRuleにバインドされたデフォルトのクラスです。
In [7]:
rr1 = ode.ODEReactionRule()
rr1.add_reactant(Species("A"))
rr1.add_reactant(Species("B"))
rr1.add_product(Species("C"))
rl1 = ode.ODERatelawMassAction(2.0)
rr1.set_ratelaw(rl1) # equivalent to rr1.set_k(2.0)
print(rr1.as_string())
A+B>C|2
ode.ODERatelawCallbackは、フラックスを計算するためのユーザ定義関数を有効にします。
In [8]:
def mass_action(reactants, products, volume, t, rr):
veloc = 2.0 * volume
for value in reactants:
veloc *= value / volume
return veloc
rl2 = ode.ODERatelawCallback(mass_action)
rr1.set_ratelaw(rl2)
print(rr1.as_string())
A+B>C|mass_action
bound関数は5つの引数を受け入れ、浮動小数点を速度として返す必要があります。
第1および第2のリストは、それぞれ反応物および生成物のそれぞれの値を含みます。
化学量論係数にアクセスする必要があるときは、引数に
rr(ode.ODEReactionRule)を使います。
ラムダ関数も利用可能です。
In [9]:
rl2 = ode.ODERatelawCallback(lambda r, p, v, t, rr: 2.0 * r[0] * r[1])
rr1.set_k(0)
rr1.set_ratelaw(rl2)
print(rr1.as_string())
A+B>C|<lambda>
6.3. ode.ODENetworkModel¶
ode.ODENetworkModelはReactionRuleと
ode.ODEReactionRuleの両方を受け入れます。
ReactionRuleは暗黙的に変換され、ode.ODEReactionRuleとして保存されます。
In [10]:
m1 = ode.ODENetworkModel()
rr1 = create_unbinding_reaction_rule(Species("C"), Species("A"), Species("B"), 3.0)
m1.add_reaction_rule(rr1)
rr2 = ode.ODEReactionRule(create_binding_reaction_rule(Species("A"), Species("B"), Species("C"), 0.0))
rr2.set_ratelaw(ode.ODERatelawCallback(lambda r, p, v, t, rr: 0.1 * r[0] * r[1]))
m1.add_reaction_rule(rr2)
ode.ODENetworkModel中のode.ODEReactionRuleのリストには、モデルのメンバー
reaction_rules()を介してアクセスできます。
In [11]:
print([rr.as_string() for rr in m1.reaction_rules()])
['C>A+B|3', 'A+B>C|<lambda>']
最後に、次のように他のソルバーと同じ方法でシミュレーションを実行できます。
In [12]:
run_simulation(1.0, model=m1, y0={'A': 60, 'B': 60})
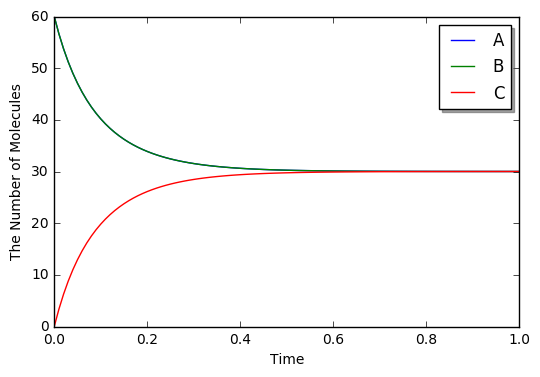
Python のデコレータを使用したモデリングは、レート(浮動小数)の代わりに関数を指定することによっても利用できます。 浮動小数点数が設定されている場合、それは質量作用反応の運動速度であると仮定されますが、一定速度ではありません。
In [13]:
with reaction_rules():
A + B == C | (lambda r, *args: 0.1 * reduce(mul, r), 3.0)
m1 = get_model()
For the simplicity, you can directory defining the equation with
Species names as follows:
In [14]:
with reaction_rules():
A + B == C | (0.1 * A * B, 3.0)
m1 = get_model()
反応物や生成物としてリストアップされていないSpeciesを(速度則中で)呼び出すと、自動的に酵素としてリストに追加されます。
In [15]:
with reaction_rules():
S > P | 1.0 * E * S / (30.0 + S)
m1 = get_model()
print(m1.reaction_rules()[0].as_string())
S+E>P+E|((1.0*E*S)/(30.0+S))
ここで式中のEは、反応物および生成物リストの両方に付加されます。
In [16]:
run_simulation(10.0, model=m1, y0={'S': 60, 'E': 30})
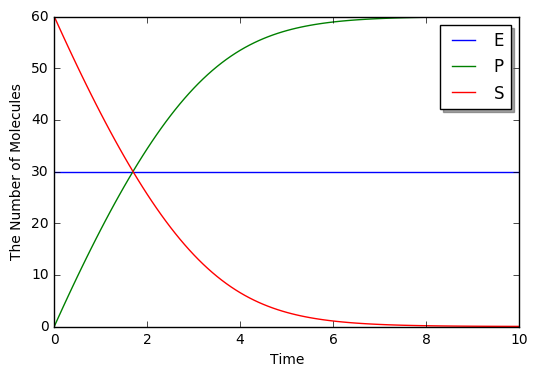
Speciesの名前の誤字に注意してください。
タイプミスをした場合、それは意図せず新しい酵素として認識されてしまいます:
In [17]:
with reaction_rules():
A13P2G > A23P2G | 1500 * A13B2G # typo: A13P2G -> A13B2G
m1 = get_model()
print(m1.reaction_rules()[0].as_string())
A13P2G+A13B2G>A23P2G+A13B2G|(1500*A13B2G)
酵素の自動宣言を無効にしたい場合は、
util.decorator.ENABLE_IMPLICIT_DECLARATIONを無効にしてください。
その値が Falseの場合、上記の場合はエラーが発生します:
In [20]:
util.decorator.ENABLE_IMPLICIT_DECLARATION = False
try:
with reaction_rules():
A13P2G > A23P2G | 1500 * A13B2G
except RuntimeError as e:
print(repr(e))
util.decorator.ENABLE_IMPLICIT_DECLARATION = True
RuntimeError('unknown variable [A13B2G] was used.',)
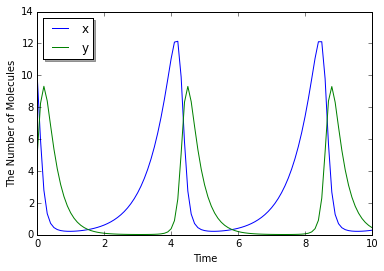
E-Cell4は、生化学反応ネットワークのシミュレーションに特化していますが、合成反応ルールを使用することで、常微分方程式を直感的に変換することができます。 たとえば、Lotka-Volterraの式は次のようになります。
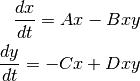
ここで 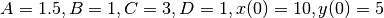 は以下のように解かれます。
は以下のように解かれます。
In [19]:
with reaction_rules():
A, B, C, D = 1.5, 1, 3, 1
~x > x | A * x - B * x * y
~y > y | -C * y + D * x * y
run_simulation(10, model=get_model(), y0={'x': 10, 'y': 5})
6.4. 速度則における参照について¶
ここで、速度則の定義の詳細を説明します。
第1に、Species
で速度則のより単純な定義を使用すると、限られた数の数学関数(例えば
exp,log,sin,cos,tan,asin,acos,atan,やpiなど)
はブロック外で関数を宣言してもそこで利用できます。
In [20]:
try:
from math import erf
with reaction_rules():
S > P | erf(S / 30.0)
except TypeError as e:
print(repr(e))
TypeError('a float is required',)
This is because erf is tried to be evaluated agaist S / 30.0
first, but it is not a floating number. In contrast, the following case
is acceptable:
In [21]:
from math import erf
with reaction_rules():
S > P | erf(2.0) * S
m1 = get_model()
print(m1.reaction_rules()[0].as_string())
S>P|(0.995322265019*S)
erf(2.0)、
0.995322265019の結果だけがレート法に渡されます。
したがって、上記のレート法は erf関数への参照を持ちません。
同様に、外部に宣言された変数の値は受け入れ可能ですが、参照としては使用できません。
In [22]:
kcat, Km = 1.0, 30.0
with reaction_rules():
S > P | kcat * E * S / (Km + S)
m1 = get_model()
print(m1.reaction_rules()[0].as_string())
kcat = 2.0
print(m1.reaction_rules()[0].as_string())
S+E>P+E|((1.0*E*S)/(30.0+S))
S+E>P+E|((1.0*E*S)/(30.0+S))
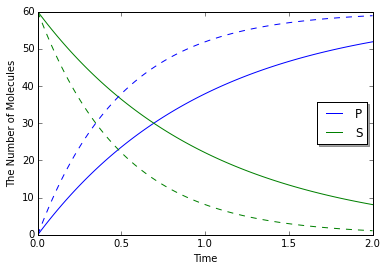
変数の値を変更しても、速度則には影響しません。 一方、独自の関数を使用して速度則を定義すると、外部の変数への参照を保持することができます。
In [23]:
k1 = 1.0
with reaction_rules():
S > P | (lambda r, *args: k1 * r[0]) # referring k1
m1 = get_model()
obs1 = run_simulation(2, model=m1, y0={"S": 60}, return_type='observer')
k1 = 2.0
obs2 = run_simulation(2, model=m1, y0={"S": 60}, return_type='observer')
viz.plot_number_observer(obs1, '-', obs2, '--')
ただし、この場合、パラメータのセットごとに新しいモデルを作成する方が良いです。
In [24]:
def create_model(k):
with reaction_rules():
S > P | k
return get_model()
obs1 = run_simulation(2, model=create_model(k=1.0), y0={"S": 60}, return_type='observer')
obs2 = run_simulation(2, model=create_model(k=2.0), y0={"S": 60}, return_type='observer')
# viz.plot_number_observer(obs1, '-', obs2, '--')
6.5. odeについてより深く¶
ode.ODEWorldでは、各Speciesの値は浮動小数です。
しかし、互換性のために、共通メンバ
num_moleculesとadd_moleculesはその値を整数とみなします。
In [25]:
w = ode.ODEWorld()
w.add_molecules(Species("A"), 2.5)
print(w.num_molecules(Species("A")))
2
実数を設定/取得するには、 set_valueとget_valueを使います:
In [26]:
w.set_value(Species("B"), 2.5)
print(w.get_value(Species("A")))
print(w.get_value(Species("B")))
2.0
2.5
デフォルトでは
ode.ODESimulatorはROSENBROCK4_CONTROLLERというRosenblock法を使用してODEを解きます。
それに加えて、2つのソルバー、
EULERとRUNGE_KUTTA_CASH_KARP54が利用可能です。
ROSENBROCK4_CONTROLLERとRUNGE_KUTTA_CASH_KARP54はエラー制御により時間発展中のステップサイズを適応的に変更しますが、
EULERはこれを行いません。
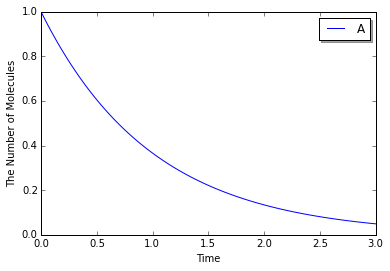
In [27]:
with reaction_rules():
A > ~A | 1.0
m1 = get_model()
w1 = ode.ODEWorld()
w1.set_value(Species("A"), 1.0)
sim1 = ode.ODESimulator(m1, w1, ode.EULER)
sim1.set_dt(0.01) # This is only effective for EULER
sim1.run(3.0, obs1)
ode.ODEFactoryは、ソルバ型とデフォルトのステップ間隔も受け入れます。
In [28]:
run_simulation(3.0, model=m1, y0={"A": 1.0}, factory=ode.ODEFactory(ode.EULER, 0.01))
下記の例も参照してください。